..enkel metod..
Typiskt pensionärsnöje ;-) och visst går det med Slackware också, det finns en "live-cd" (baserad på Ubuntu) här..
https://www.gramps-project.org/wiki/index.php?title=Linux_Genealogy_CD
..om du vill titta på lite olika.
Jag har valt GeneWeb och PhpGedView
Det finns ett mycket bra program som heter Gramps vilket fungerar utmärkt om du använder någon "Gnome-Slackware", själv tog jag en titt på "beroendelistan" och insåg direkt att jag hade roligare saker att göra. ;-)
Alla andra släktingar till mig är ju typiskt "Windowsanpassade", därav detta program som relativt enkelt går att installera där också, naturligtvis skrev jag en guide om det också :-)
(https://web.comhem.se/haze2/windows/geneweb/geneweb.html) GeneWeb på Windows
Som vanligt ska man alltid läsa åtminstone README och INSTALL när man packat upp ett program, jag skriver det nu så slipper ni höra detta "tjat" varje gång. ;-)
Du behöver "Objective Caml compiler"..
https://caml.inria.fr/ocaml/release.en.html
..för närvarande 3.09.3, jag tar "tar.gz", jag vill ha sådana program i /usr/local så jag packar upp där (som "root")
Se till att du är root i /usr/local..
bash-3.2$ su
Password:
bash-3.2# cd /usr/local
..packa upp
bash-3.2# tar xvfz /där/du/laddat/ner/ocaml-3.09.3.tar.gz
flytta till programkatalogen..
bash-3.2# cd ocaml-3.09.3/
..konfigurera..
bash-3.2# ./configure
..bla..bla..
** Configuration summary **
Directories where Objective Caml will be installed:
binaries.................. /usr/local/bin
standard library.......... /usr/local/lib/ocaml
manual pages.............. /usr/local/man (with extension .1)
Configuration for the bytecode compiler:
C compiler used........... gcc
options for compiling..... -fno-defer-pop -Wall -D_FILE_OFFSET_BITS=64 -D_REENTRANT
options for linking....... -Wl,-E -lm -ldl -lcurses -lpthread
shared libraries are supported
options for compiling..... -fPIC -fno-defer-pop -Wall -D_FILE_OFFSET_BITS=64 -D_REENTRANT
command for building...... gcc -shared -o lib.so -Wl,-rpath,/a/path objs
Configuration for the native-code compiler:
hardware architecture..... i386
OS variant................ linux_elf
C compiler used........... gcc
options for compiling..... -Wall -D_FILE_OFFSET_BITS=64 -D_REENTRANT
options for linking....... -lm
assembler ................ $(AS)
preprocessed assembler ... gcc -c -DSYS_$(SYSTEM)
profiling with gprof ..... supported
Source-level replay debugger: supported
Additional libraries supported:
unix str num dynlink bigarray systhreads threads graph labltk
Configuration for the "num" library:
target architecture ...... ia32 (asm level 2)
Configuration for the "graph" library:
options for compiling .... -I/usr/X11R6/include
options for linking ...... -L/usr/X11R6/lib -lX11
Configuration for the "labltk" library:
use tcl/tk version ....... 8.4
options for compiling ....
options for linking ...... -ltk8.4 -ltcl8.4 -ldl
** Objective Caml configuration completed successfully **
..bygg först..
bash-3.2# make world
..testa..
bash-3.2# make bootstrap
..bla..bla..
make compare
make[1]: Entering directory `/usr/local/ocaml-3.09.3'
Fixpoint reached, bootstrap succeeded.
make[1]: Leaving directory `/usr/local/ocaml-3.09.3'
..bygg slutligen
bash-3.2# make opt
..installera..
bash-3.2# make install
Ladda ner senaste stabila versionen av GeneWeb..
https://cristal.inria.fr/~ddr/GeneWeb/ftp/Linux/
..för närvarande gw-5.00-ln26-x86.tgz
Se till att du är vanlig användare i den katalog där du laddade ner programmet..
..packa upp..
bash-3.2$ tar xvfz /där/du/laddat/ner/geneweb-5.00.tgz
flytta till programkatalogen..
bash-3.2$ cd geneweb-5.00
..konfigurera (detta går snabbt)..
bash-3.2# ./configure
..enl INSTALL..
If the command ocamlc.opt is not installed, edit the file
"tools/Makefile.inc" and change:
OCAMLC=ocamlc.opt
OCAMLOPT=ocamlopt.opt
into
OCAMLC=ocamlc
OCAMLOPT=ocamlopt
Vi har inte "ocamlc.opt" så vi ändrar och sedan..
bash-3.2# make
..följt av..
bash-3.2# make distrib
Nu har du en katalog "distribution" som innehåller allt. :-).
Jag kopierade min till /home/titus och ändrade namnet till geneweb500
bash-3.2# cp -r distribution/ /home/titus/geneweb500
..flytta dit..
bash-3.2# cd /home/titus/geneweb500
Nu ska vi ställa in programmet..
bash-3.2# ./gwsetup
* Deutsche Version: "de" eingeben
* English version: type "en"
* Version espanola: escribir en el teclado "es"
* Suomalainen versio: kirjoita "fi"
* Version francaise: tapez "fr"
* Versione italiana: digitate "it"
* Latvieðu versija: type "lv"
* Svensk version: skriv "sv"
? sv
..vi ska ha svenska så sv och tryck "enter"
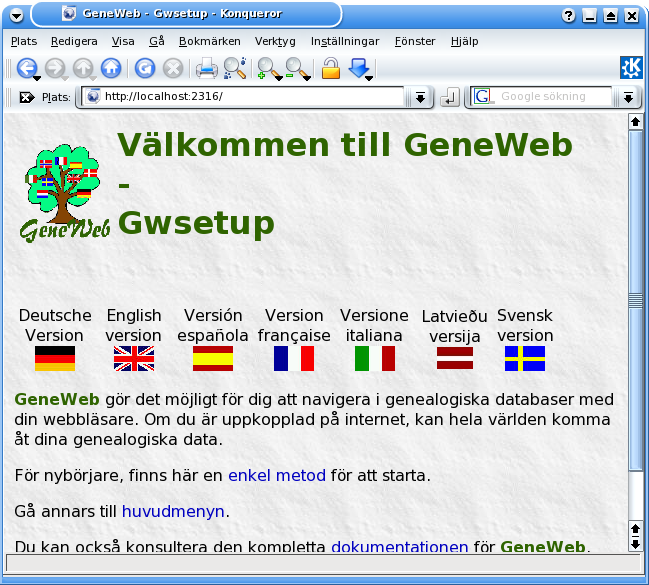
Välkommen till GeneWeb 5.00
[Go to your Web browser and open one of the following addresses:]
https://127.0.0.1:2316/
https://localhost:2316/
Varning: för att det här programmet ska fungera, får du inte stänga
detta fönster!
Om det inte fungerar, kanske du måste ställa in din webbläsare
med optioner som "arbeta off-line" eller "använd inte en proxyserver
för lokala adresser" eller "anslut inte till Internet för adressen
'localhost'".
Ready 2007-04-13 13:47 port 2316...
..jag öppnar med "Konqueror"..

..enkel metod..
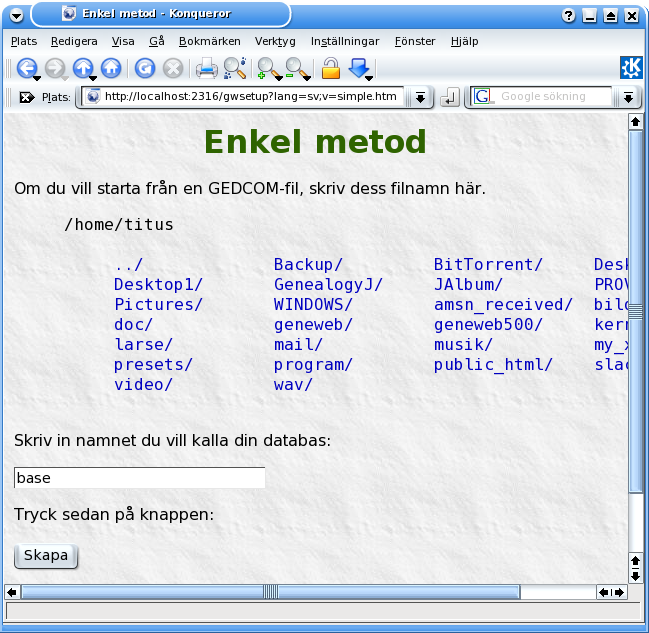
..kalla den vad du vill..
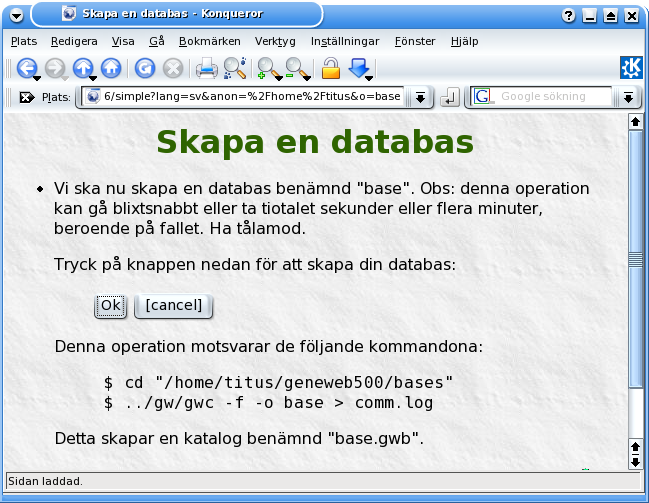
..påminnelse..
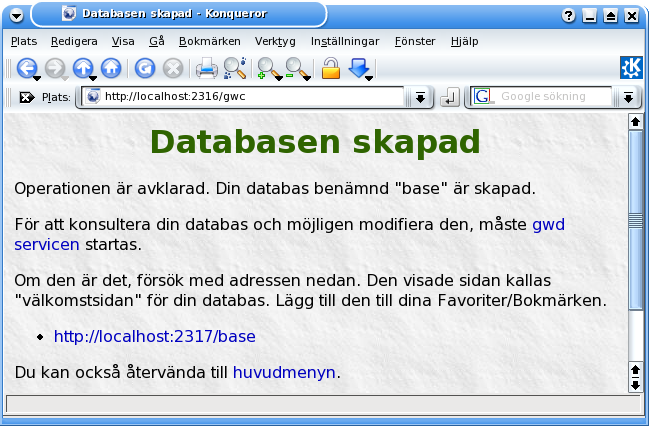
..nu kan du via "ctrl+c" stänga din terminal så får du prompten tillbaka.Det finns
ett skript i huvudkatalogen (geneweb500) som enkelt startar "gwd-servicen"
Ställ dig där och.. bash-3.2# ./gwd GeneWeb 5.00 - Copyright (c) 1998-2007 INRIA Possible addresses: https://localhost:2317/base https://127.0.0.1:2317/base https://zenita:2317/base where "base" is the name of the database Type control C to stop the service Ready 2007-04-13 14:07 port 2317... Detta fönster måste var öppet så länge du använder programmet. Då tar jag den nya adressen i "Konkan"..
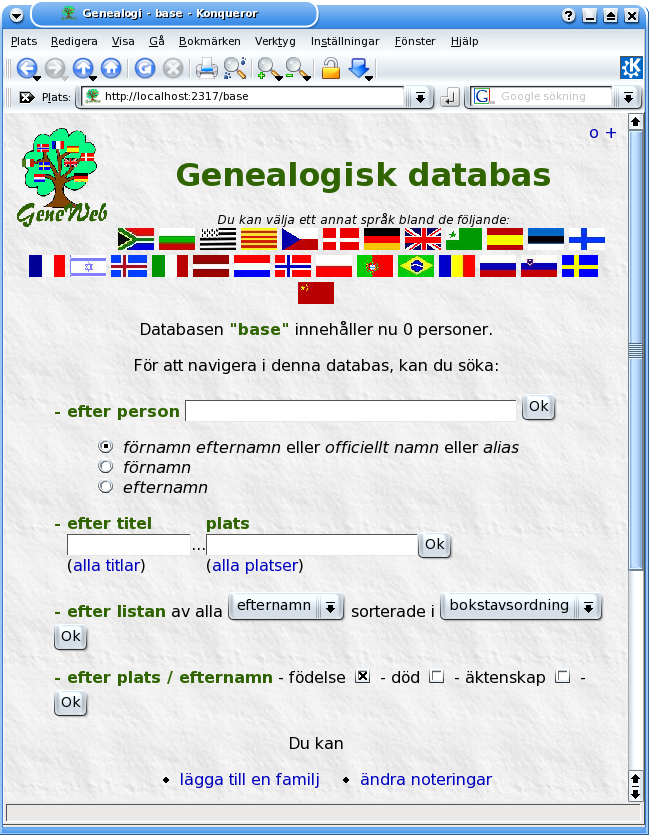
..databasen är tom,så börja med att lägga till en Familj..
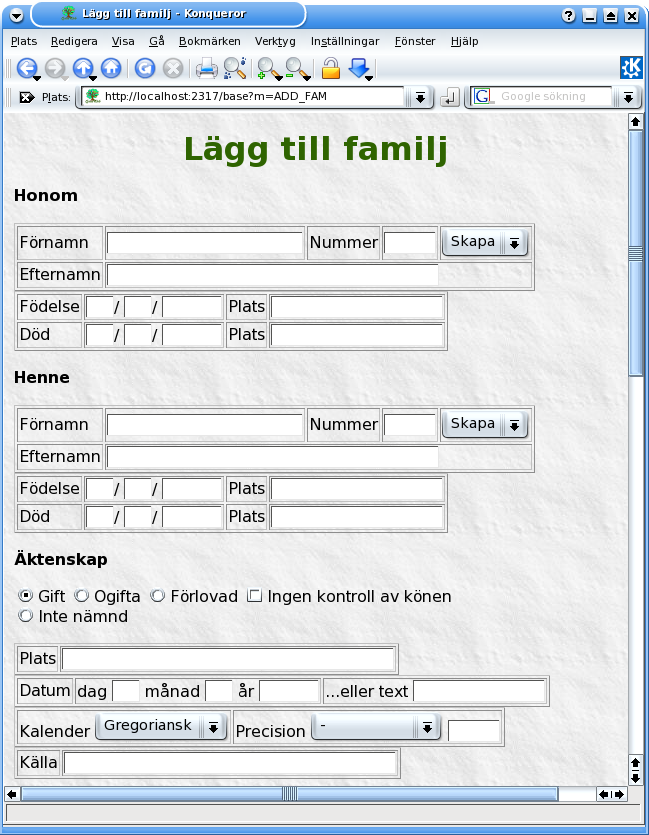
..så är du igång! All dokumentation/manual har du i katalogen gw/doc t.o.m på Svenska. :-) Det här programmet kan du också ha på "Webben", läs mer om det i dokumentationen, men jag tycker PhpGedView är bra mycket bättre så då visar jag det istället.
PhpGedView